サブグループ解析の結果をフォレストプロットで書く方法はいくつかあるが、ここでは、R スクリプト中心に書く方法を解説する
EZR の機能を使う方法
R のスクリプトの方法の前に、EZR のメニューをいろいろ駆使して書く方法の記事をご紹介
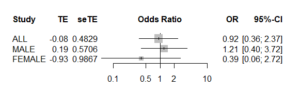
EZR になれている場合、こちらのほうがよいかもしれない
R スクリプトでサブグループ解析結果をフォレストプロットで書く方法
R スクリプトで書く場合は、以下のように実行する
3 ステップに分けて説明する
ここでは、EZR 公式マニュアル付属のデータセットを活用して、Cox 回帰のサブグループ解析を行う
サブグループ解析結果の前に全体解析結果をデータフレームに格納する
サブグループ解析の結果をデータフレームに格納していくのだが、その前に、全体解析の結果の対数ハザード比 logHR と標準誤差 SE をデータフレームに格納する
# Forest plot of subgroup analysis
# サブグループごとの解析を行い結果を df に格納する
load("ElderlyAML_rev.RData")
library(survival)
df <- data.frame()
res <- coxph(Surv(Days,Survival)~Anthracyclines, data=ElderlyAML)
logHR <- summary(res)$coef[1]
SE <- summary(res)$coef[3]
GR <- 'ALL'
res1 <- data.frame(GR, logHR, SE)
df <- rbind(df,res1)
dfデータフレーム df の先頭行に ALL の結果を格納した
> df
GR logHR SE
1 ALL -0.9215332 0.3455331サブグループ解析の結果をデータフレームに格納する
次に、サブグループ解析の結果をデータフレームに格納する
variables にサブグループ解析を行うカテゴリカルデータの変数名ベクトルを格納して、使用する
variables <- c('Sex.Dummy.F', 'PS2', 'LDH250', 'Cre15', 'CRP1', 'WBC30000', 'Age70', 'Plt2')
for (variable in variables){
res <- coxph(Surv(Days,Survival) ~ Anthracyclines, data = ElderlyAML, subset = get(variable) == 0)
logHR <- summary(res)$coef[1]
SE <- summary(res)$coef[3]
GR <- paste(variable, "=0", sep="")
res1 <- data.frame(GR, logHR, SE)
df <- rbind(df, res1)
res <- coxph(Surv(Days,Survival) ~ Anthracyclines, data = ElderlyAML, subset = get(variable) == 1)
logHR <- summary(res)$coef[1]
SE <- summary(res)$coef[3]
GR <- paste(variable, "=1", sep="")
res1 <- data.frame(GR, logHR, SE)
df <- rbind(df, res1)
}
dfvariables の一つ一つのカテゴリカルデータの 0 のときの結果と、1 のときの結果を順番にデータフレームに格納している
できあがりは、以下のとおりである
> df
GR logHR SE
1 ALL -0.9215332 0.3455331
2 Sex.Dummy.F=0 -0.8433599 0.4774421
3 Sex.Dummy.F=1 -1.0185934 0.5133041
4 PS2=0 -0.9211330 0.3780901
5 PS2=1 -0.5377643 0.8248212
6 LDH250=0 -1.7529075 0.8527541
7 LDH250=1 -0.7791516 0.3971939
8 Cre15=0 -0.8331148 0.3816948
9 Cre15=1 -1.3697384 1.1391895
10 CRP1=0 -1.0266389 0.5534791
11 CRP1=1 -0.8795466 0.4542963
12 WBC30000=0 -1.0448020 0.4100158
13 WBC30000=1 -0.6614039 0.6758000
14 Age70=0 -1.4822192 0.5713317
15 Age70=1 -0.2119710 0.4554232
16 Plt2=0 -1.0395092 0.3858093
17 Plt2=1 -0.7244348 0.9243423メタアナリシスパッケージの関数を借りてフォレストプロットを書く
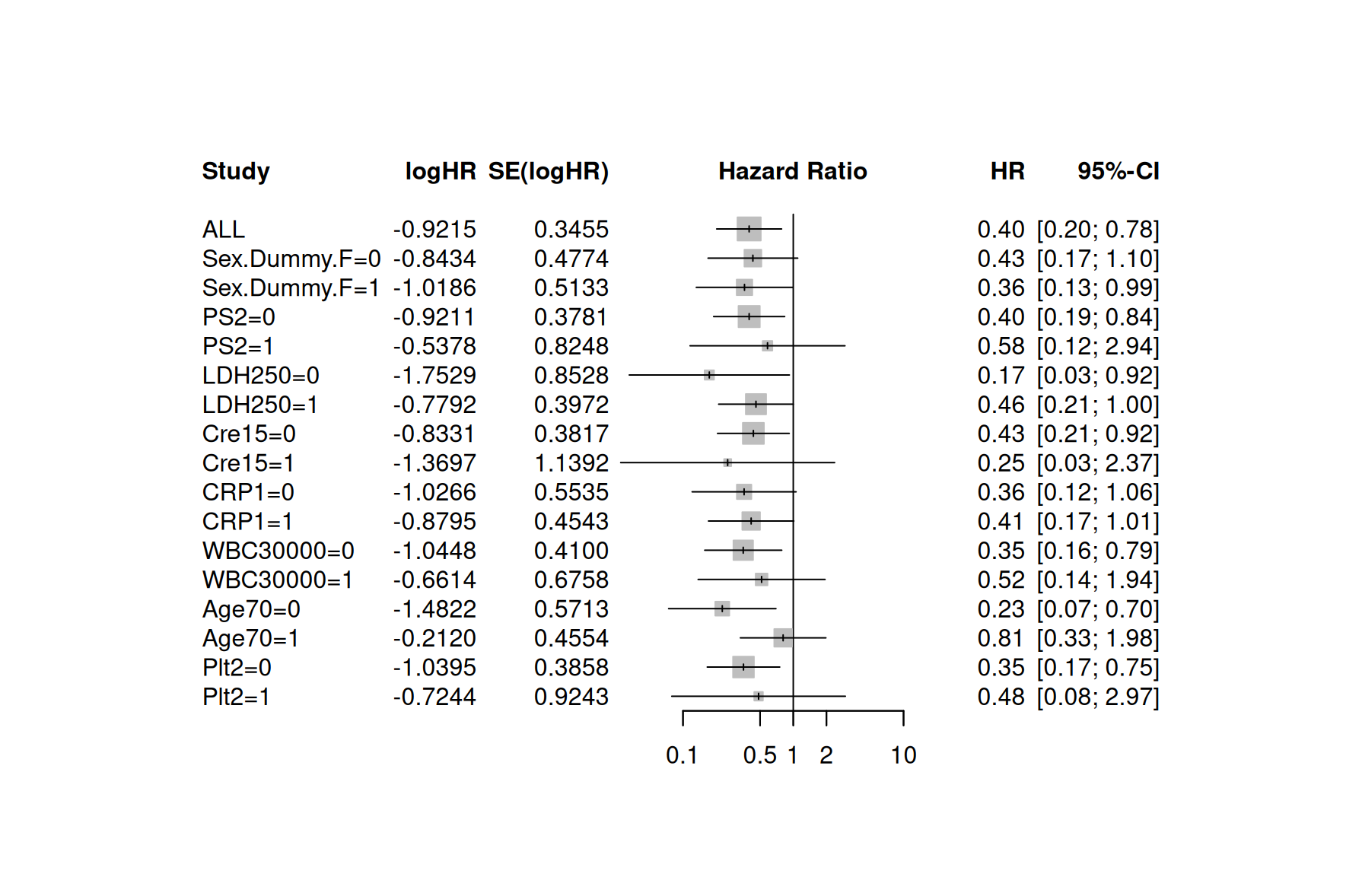
メタアナリシスパッケージの meta にある、metagen 関数と forest 関数を借りて、フォレストプロットを書く
# メタアナリシスの関数を借りて、フォレストプロットを書く
library(meta)
metagen.res <- metagen(TE=logHR, seTE=SE, studlab=GR, data=df, sm='HR', common=FALSE, random=FALSE)
forest(metagen.res)以下のように、メタアナリシスのフォレストプロットと同様のグラフが、サブグループ解析結果として並んで書かれる
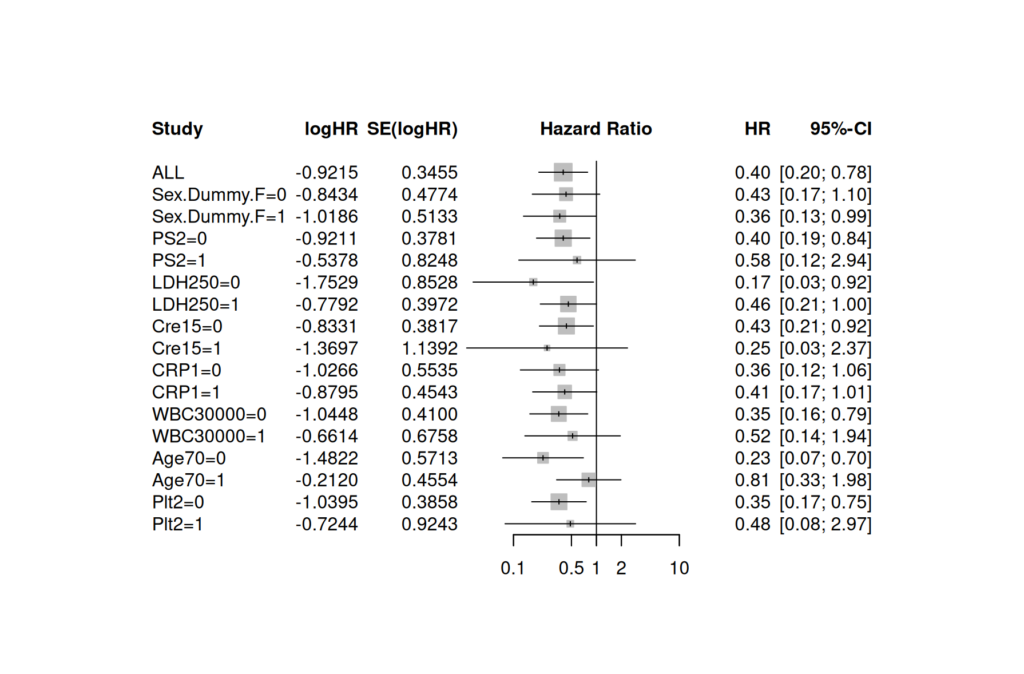
まとめ
R でサブグループ解析結果をフォレストプロットで書く方法を紹介した
R スクリプトで、二値のカテゴリカルデータによる複数のサブグループを、for 文で一気に計算して、最後に metagen と forest で書くという流れである
参考になれば





コメント