Rでカプランマイヤー曲線を書く方法の紹介。
survfit を使ったグループごとの曲線の書き方。
Rでカプランマイヤー曲線を書くためのサンプルデータ
カプランマイヤー曲線を書くためのサンプルデータは、survival パッケージの lung を使う。
これは、North Central Cancer Treatment Group, NCCTG 提供の進行肺がん患者さんの生存データ。
まず、survival パッケージの関数及びサンプルデータが使えるようにする。
library(survival)
head()で先頭部分を見てみると以下の通り。timeが生存時間(日数)、statusが打ち切り(1)か死亡(2)か、ph.ecogがECOG performance scoreでgood(0)からdead(5)までのグレーディング。
> head(lung)
inst time status age sex ph.ecog ph.karno pat.karno meal.cal wt.loss
1 3 306 2 74 1 1 90 100 1175 NA
2 3 455 2 68 1 0 90 90 1225 15
3 3 1010 1 56 1 0 90 90 NA 15
4 5 210 2 57 1 1 90 60 1150 11
5 1 883 2 60 1 0 100 90 NA 0
6 12 1022 1 74 1 1 50 80 513 0
Rでカプランマイヤー曲線を書くための準備その1:Surv()
Surv()は、生存時間とステータスを使って、生存時間データに変換してくれる関数。
> with(data=lung, Surv(time, status))[1:20]
[1] 306 455 1010+ 210 883 1022+ 310 361 218 166 170 654
[13] 728 71 567 144 613 707 61 88
先頭の20個を取り出してみると上記のように変換されている。1010と1022に+がついているのは、打ち切り例であることを示している。
Rでカプランマイヤー曲線を書くための準備その2:survfit()
survfit()は、Surv()で生存時間データに変換された数値をもとに、生存時間の推定計算をする。
> survfit(Surv(time, status)~1, data=lung)
Call: survfit(formula = Surv(time, status) ~ 1, data = lung)
n events median 0.95LCL 0.95UCL
228 165 310 285 363
サンプルサイズが228、イベントが165、生存期間中央値が310日、95%信頼区間が285日から363日と計算されている。
Rでカプランマイヤー曲線を書いてみる:plot()
survfit()に何か名前を付けて(ここでは survfit1 が付けた名前)、その名前をplot()に入れるだけで、カプランマイヤー曲線が書ける。
survfit1 <- survfit(Surv(time, status)~1, data=lung)
plot(survfit1)
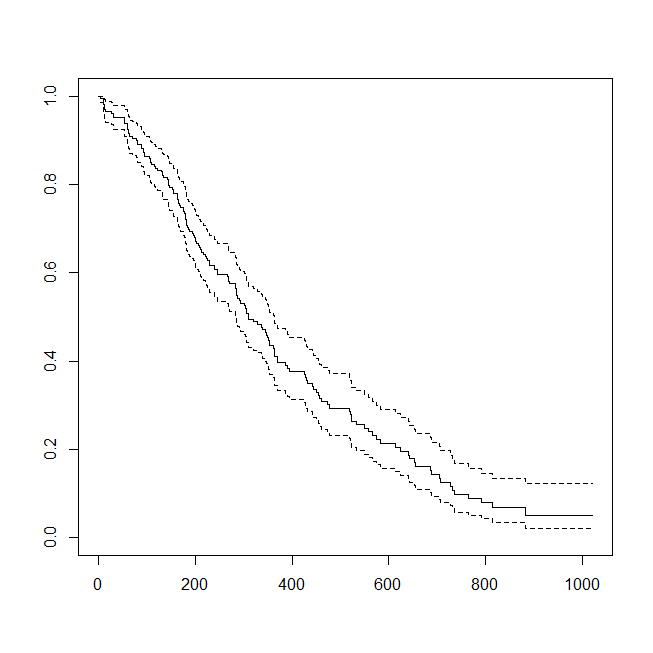
実線の上下の破線は95%信頼区間を示している。表示を消すにはconf.int=FALSEを追加する。
plot(survfit1, conf.int=FALSE)
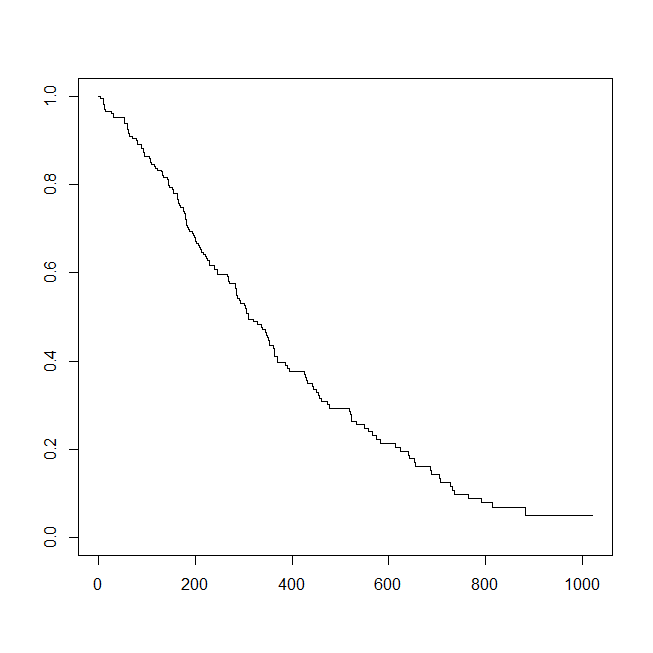
Y軸の目盛り数値を縦にするには las=1、X軸とY軸の説明は xlab=””, ylab=””で書き入れる。
plot(survfit1, conf.int=FALSE, las=1,
xlab="Survival Time (days)", ylab="Overall Survival")
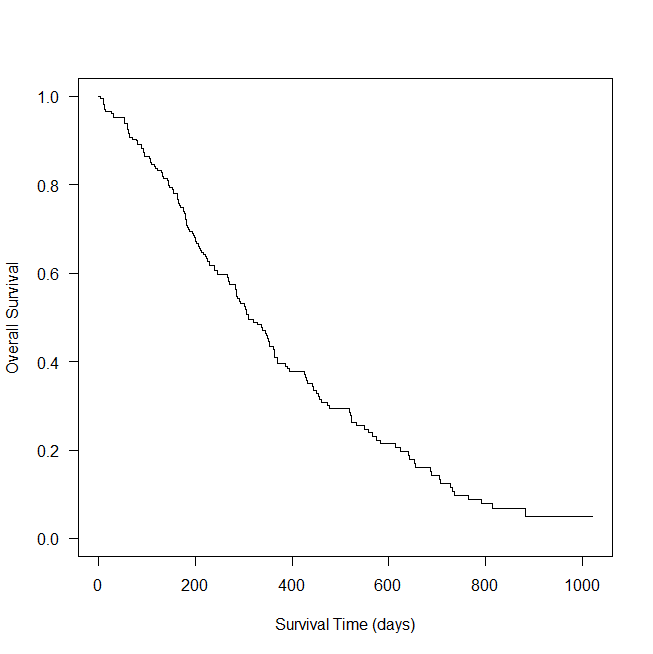
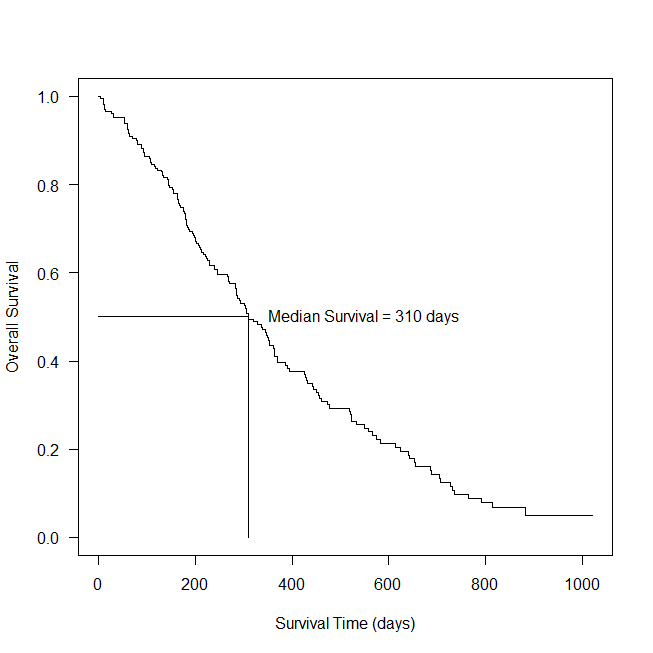
生存時間中央値を図に書き入れるには、arrows()を使う。始点と終点を座標で示して、矢印の大きさをゼロにする。
また、図中にコメントを入れるには text()を使う。指定したX座標が、テキストの中点になる。
arrows(310,0,310,0.5,0)
arrows(0,0.5,310,0.5,0)
text(550,0.5,"Median Survival = 310 days")
Rでカプランマイヤー曲線をグループごとに書くには?
カプランマイヤー曲線をいくつかのグループに分けて書くにはどうしたらよいか?
グループに分けるには survfit() で「~」の後に、グループわけの変数を入れる。
ECOGのパフォーマンススコアで分けて書くなら、~ph.ecogと書く。
survfit2 <- survfit(Surv(time,status)~ph.ecog, data=lung)
まず、survfit2を確認してみると、ECOG Performance Scoreは0から3までの患者さんしかいなかった。また、3の人は一人のみであった。
> survfit2
Call: survfit(formula = Surv(time, status) ~ ph.ecog, data = lung)
1 observation deleted due to missingness
n events median 0.95LCL 0.95UCL
ph.ecog=0 63 37 394 348 574
ph.ecog=1 113 82 306 268 429
ph.ecog=2 50 44 199 156 288
ph.ecog=3 1 1 118 NA NA
plot()で作図する。legend()で、図中に注釈を入れる。
plot(survfit2,las=1,xlab="Survival Time (days)",
ylab="Overall Survivial",col=2:5)
legend("topright", paste("ECOG PS=",0:3, sep=""), lty=1,
col=2:5)
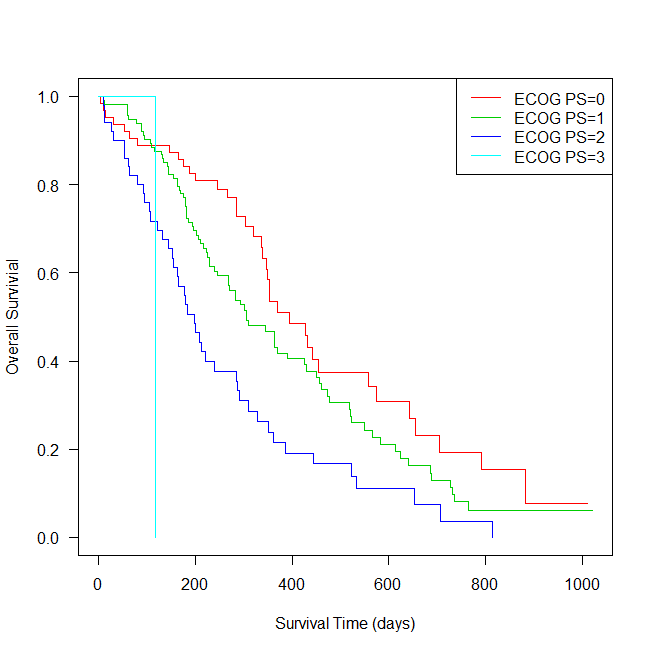
これで、グループ別のカプランマイヤー曲線が書けた。
凡例内の記載をデータから持ってくることもできる。legend=, names(), $strata を使って以下のように書ける。
plot(survfit2,las=1, xlab="Survival Time (days)",
ylab="Overall Survivial",col=2:5)
legend("topright", legend=names(survfit2$strata), lty=1,
col=2:5)
グラフはこのようになる。
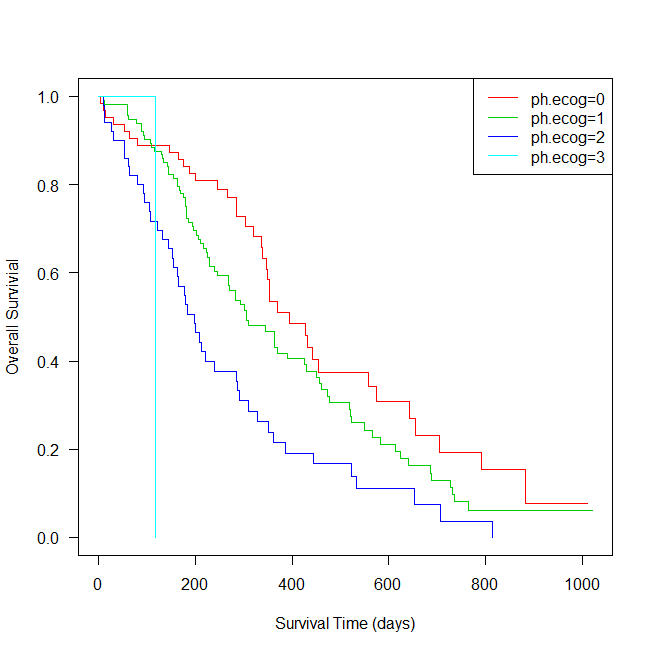
まとめ
Rでカプランマイヤー曲線を書く方法を紹介した。
survival パッケージを呼び出して、Surv(), survfit(), plot()の3つで基本は書ける。
グループごとに書かせるためには、survfit()中に「~グループ名」を入れる。
plot()のカッコ内に las=, xlab=, ylab= を使うと最低限の見栄えのグラフにはなる。
必要に応じて、arrows(), text(), legend()でさらにきれいなグラフを描くことも可能。
参考になれば。
関連記事





コメント
コメント一覧 (3件)
[…] […]
[…] […]
[…] […]