分割プロット分散分析 split-plot design は、群分けのある反復測定分散分析のこと
分割プロット分散分析を R で行う方法。
分割プロット分散分析のサンプルデータ
今回使っていくサンプルデータはこちら。
X1_0 <- c(7.5,10.6,12.4,11.5,8.3,9.2)
X1_1 <- c(8.6,11.7,13.0,12.6,8.9,10.1)
X1_2 <- c(6.9,8.8,11.0,11.1,6.8,8.6)
X1_3 <- c(0.8,1.6,5.6,7.5,0.5,3.8)
X2_0 <- c(13.3,10.7,12.5,8.4,9.4,11.3)
X2_1 <- c(13.3,10.8,12.7,8.7,9.6,11.7)
X2_2 <- c(12.9,10.7,12.0,8.1,8.0,10.0)
X2_3 <- c(11.1,9.3,10.1,5.7,3.8,8.5)
dat1 <- data.frame(X1_0,X1_1,X1_2,X1_3)
dat2 <- data.frame(X2_0,X2_1,X2_2,X2_3)
データをグラフにしてみる
グラフ描画のスクリプトはこちら。
matplot(t(dat1),las=1,xlab="Week",ylab="Measurement",type="b",
xaxt="n", main="Group 1",ylim=c(0,15))
axis(side=1,at=1:4,label=0:3)
matplot(t(dat2),las=1,xlab="Week",ylab="Measurement",type="b",
xaxt="n", main="Group 2",ylim=c(0,15))
axis(side=1,at=1:4,label=0:3)
matplot()で行列のデータをプロットする。
グラフ中の数字は症例番号で、1番さん、2番さん、・・・ ということになる。
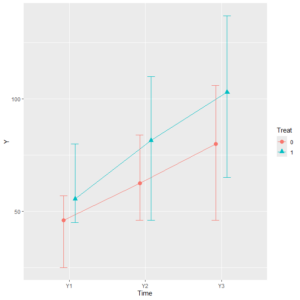
グラフはこんな感じ。
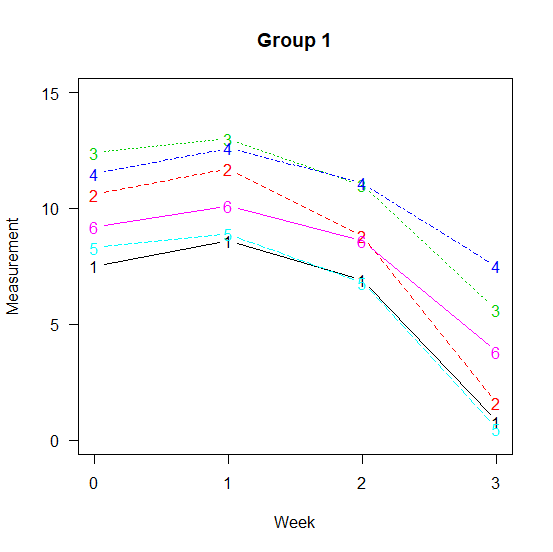
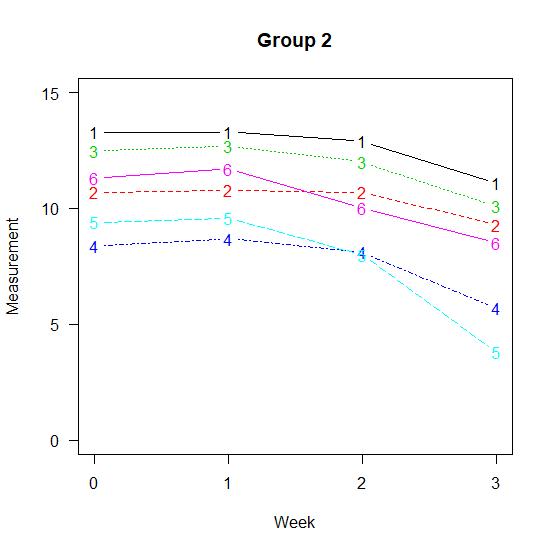
Group2に比べるとGroup1のほうが測定値が大きく減少している傾向が見える。
分割プロット分散分析用にデータを並べ替えて解析しやすくする
解析用にデータを並べ替える。
X <- c(X1_0,X1_1,X1_2,X1_3,X2_0,X2_1,X2_2,X2_3)
A <- factor(c(rep(1,6*4),rep(2,6*4)))
T <- factor(c(sort(rep(0:3,6)),sort(rep(0:3,6))))
R <- factor(c(rep(1:6,8)))
data.frame(X,A,T,R)
Xが測定値、Aが要因でGroup1とGroup2、Tが測定回で、0がベースライン、1,2,3はそれぞれ1回目、2回目、3回目。
RはAのGroupごとの症例番号(例題はラットなので実験動物番号)。
どちらのGroupも6番までいる。
データを見てみるとこんな感じ。
> data.frame(X,A,T,R)
X A T R
1 7.5 1 0 1
2 10.6 1 0 2
3 12.4 1 0 3
4 11.5 1 0 4
5 8.3 1 0 5
6 9.2 1 0 6
7 8.6 1 1 1
8 11.7 1 1 2
9 13.0 1 1 3
10 12.6 1 1 4
11 8.9 1 1 5
12 10.1 1 1 6
13 6.9 1 2 1
14 8.8 1 2 2
15 11.0 1 2 3
16 11.1 1 2 4
17 6.8 1 2 5
18 8.6 1 2 6
19 0.8 1 3 1
20 1.6 1 3 2
21 5.6 1 3 3
22 7.5 1 3 4
23 0.5 1 3 5
24 3.8 1 3 6
25 13.3 2 0 1
26 10.7 2 0 2
27 12.5 2 0 3
28 8.4 2 0 4
29 9.4 2 0 5
30 11.3 2 0 6
31 13.3 2 1 1
32 10.8 2 1 2
33 12.7 2 1 3
34 8.7 2 1 4
35 9.6 2 1 5
36 11.7 2 1 6
37 12.9 2 2 1
38 10.7 2 2 2
39 12.0 2 2 3
40 8.1 2 2 4
41 8.0 2 2 5
42 10.0 2 2 6
43 11.1 2 3 1
44 9.3 2 3 2
45 10.1 2 3 3
46 5.7 2 3 4
47 3.8 2 3 5
48 8.5 2 3 6
分割プロット分散分析の計算 step by step
R スクリプトは以下の通り。
split.plot <- function(X,A,R,T){
Xbar <- mean(X)
Xikbar <- tapply(X, list(A,T), mean)
Xibar <- tapply(X, A, mean)
Xkbar <- tapply(X, T, mean)
a <- length(table(A))
t <- length(table(T))
r <- length(table(R))
N <- a*r
SS1 <- matrix(0,a,t)
for (i in 1:a){
for (k in 1:t){
SS1[i,k] <- r*(Xikbar[i,k]-Xibar[i]-Xkbar[k]+Xbar)^2
}
}
SS1 <- sum(SS1)
DF1 <- (a-1)*(t-1)
MS1 <- SS1/DF1
Xijbar <- tapply(X, list(A,R), mean)
SS2 <- tapply(rep(0,length(X)), list(R,T,A), mean)
Xmat <- tapply(X, list(R,T,A), mean)
for (i in 1:a){
for (j in 1:r){
for (k in 1:t){
SS2[j,k,i] <- (Xmat[j,k,i]-Xijbar[i,j]-Xikbar[i,k]+Xibar[i])^2
}
}
}
SS2 <- sum(SS2)
DF2 <- (N-a)*(t-1)
MS2 <- SS2/DF2
F <- MS1/MS2
pF <- pf(F, DF1, DF2, lower.tail=FALSE)
pF_con <- pf(F, DF1/(t-1), DF2/(t-1), lower.tail=FALSE)
list(c("SS_T*A"=SS1, "df_T*A"=DF1, "MS_T*A"=MS1, F_value=F, p_value=pF,
"SS_T*E"=SS2, "df_T*E"=DF2, "MS_T*E"=MS2, "p_conserv."=pF_con))
}
split.plot1 <- unlist(split.plot(X=X,A=A,R=R,T=T))
split.plot1
SS1とSS2の計算がキーとなる。
SS1は、時点と群の交互作用を検討する平方和。
SS2は、時点と群内誤差の平方和。
SS1とSS2の平均平方和同士の比がF値になり、群と時点の交互作用が検討できる。
結果は以下の通り。
> split.plot1
SS_T*A df_T*A MS_T*A F_value p_value
3.550000e+01 3.000000e+00 1.183333e+01 1.966759e+01 3.050506e-07
SS_T*E df_T*E MS_T*E p_conserv.
1.805000e+01 3.000000e+01 6.016667e-01 1.264629e-03
時点と群の交互作用は、F値19.67、p値が0.001未満で統計学的に有意で交互作用あり。
つまり、群によって時点での効果が違い、全体的なパターンが異なると言える。
グラフを見てもそのような傾向が見られた。
分割プロット分散分析を lme4 パッケージで実行してみる
まず、lme4パッケージをインストールする。
install.pacakges(lme4)
lme4パッケージは変量効果を含む混合モデルが実現できるパッケージ。
lme4パッケージの結果の統計学的有意性検定をするためには、さらに lmerTest パッケージのインストールも必要。
install.packages(lmerTest)
分割プロット分散分析を lme4 で行うためデータの準備
例題はグループごとに6匹のラットを使っているので、12匹のラットだ。
全部が区別できるように番号を振りなおす。
R <- factor(c(rep(1:6,8)))
R2 <- factor(c(rep(1:6,4),rep(1:6,4)+6))
dat <- data.frame(X,A,R=R2,T)
分割プロット分散分析を lme4 で行う場合 lmer() を使う
library(lme4)
library(lmerTest)
# lm01 は分割プロット分散分析を線形混合モデルで実行
lmer01 <- lmer(X ~ A*T + (1|R), data=dat)
anova(lmer01)
# lm02 は 3 元配置分散分析
lm02 <- lm(X ~ A*T*R, data=dat)
anova(lm02)
結果は以下の通り。
> anova(lmer01)
Type III Analysis of Variance Table with Satterthwaite's method
Sum Sq Mean Sq NumDF DenDF F value Pr(>F)
A 1.528 1.528 1 10 2.5404 0.142
T 205.150 68.383 3 30 113.6565 < 2.2e-16 ***
A:T 35.500 11.833 3 30 19.6676 3.051e-07 ***
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
> anova(lm02)
Analysis of Variance Table
Response: X
Df Sum Sq Mean Sq F value Pr(>F)
A 1 42.563 42.563
T 3 205.150 68.383
R 10 167.543 16.754
A:T 3 35.500 11.833
T:R 30 18.050 0.602
Residuals 0 0.000
Warning message:
In anova.lm(lm02) :
ANOVA F-tests on an essentially perfect fit are unreliable
lmer01の結果が、群と時点の交互作用を含むモデルで、ラット6匹ずつは変量効果として扱った場合。
lm02は、群と時点とラットの個体すべての固定効果と交互作用を考慮したモデル。
lmer01の分散分析結果で、時点と群の交互作用(A:T)の平方和(Sum Sq)がSS1と一致しているのがわかる。
F値とp値も上記と一致している。
lm02の分散分析結果では、時点と群内誤差の交互作用(T:R)の平方和(Sum Sq)がSS2と一致しているのがわかる。
こちらの結果は検定結果が出力されなかった。
被験者内相関を加味した方法
これまでの計算は、繰り返し測定について、同じ個体による相関を加味していない解析だ。
相関を加味した解析にはGreenhouse-Geisserの方法やHuynh-Feldtの方法がある。
これらの被験者内相関を加味した解析を簡単に実現できるのが、「ANOVA君」だ。
分割プロット分散分析に ANOVA 君を使う
ANOVA君の配布URLはこちら。
https://riseki.cloudfree.jp/?ANOVA%E5%90%9B
anovakun_???.txt を source()で読み込む。
??? には、バージョン番号が入る
記事作成時は、4.8.2 (482) であった
読み込む方法は、R consoleのメニューバーの「ファイル」→「Rコードのソースを読み込み…」でanovakun_482.txtを選択する。
以下の「happy」とか「Downloads」とかは、あなたの環境に合わせた名称に変わるはずだ。
source("C:\\Users\\happy\\Downloads\\anovakun_482.txt")
分割プロット分散分析を ANOVA 君で行うためのデータ準備
分割プロット分散分析を ANOVA 君で使えるようにデータを少し加工する。
A <- c(rep("A1",6),rep("A2",6))
X.rep <- rbind(as.matrix(dat1),as.matrix(dat2))
dat.AsB <- data.frame(A,X.rep)
結果は、このようになる。
> dat.AsB
A X1_0 X1_1 X1_2 X1_3
1 A1 7.5 8.6 6.9 0.8
2 A1 10.6 11.7 8.8 1.6
3 A1 12.4 13.0 11.0 5.6
4 A1 11.5 12.6 11.1 7.5
5 A1 8.3 8.9 6.8 0.5
6 A1 9.2 10.1 8.6 3.8
7 A2 13.3 13.3 12.9 11.1
8 A2 10.7 10.8 10.7 9.3
9 A2 12.5 12.7 12.0 10.1
10 A2 8.4 8.7 8.1 5.7
11 A2 9.4 9.6 8.0 3.8
12 A2 11.3 11.7 10.0 8.5
ANOVA君は、同じ人を一行に、繰り返し測定値は行の方向に並べるのが決まりだ。
要因Aのグループ1とグループ2だけがわかる形になっている。
この形を「AsB」と呼んでいる。
分割プロット分散分析において自由度調整ありなしでどう変わるか?
Greenhouse-Geisserの方法やHuynh-Feldtの方法は、F値を使った検定の際に、自由度を調整するというもの。
それらの自由度調整が結果にどのように影響するかがポイントだ。
gg=TRUEがGreenhouse-Geisserの方法、hf=TRUEがHuynh-Feldtの方法。
Huynh-Feldtの方法は正確には、Huynh-Feldt-Lecoutreの方法という。
Huynh-Feldtの方法が発表された後に、Lecoutreが修正式を発表し、そちらが採用されている。
anovakun(dat.AsB, "AsB", 2, 4, nopost=TRUE)
anovakun(dat.AsB, "AsB", 2, 4, nopost=TRUE, gg=TRUE)
anovakun(dat.AsB, "AsB", 2, 4, nopost=TRUE, hf=TRUE)
自由度調整なしの結果はこちら。
<< ANOVA TABLE >>
------------------------------------------------------------
Source SS df MS F-ratio p-value
------------------------------------------------------------
A 42.5633 1 42.5633 2.5404 0.1420 ns
s x A 167.5433 10 16.7543
------------------------------------------------------------
B 205.1500 3 68.3833 113.6565 0.0000 ***
A x B 35.5000 3 11.8333 19.6676 0.0000 ***
s x A x B 18.0500 30 0.6017
------------------------------------------------------------
Total 468.8067 47 9.9746
+p < .10, *p < .05, **p < .01, ***p < .001
Greenhouse-Geisserの方法による結果はこちら。
dfが小数点以下を含む数値になっていて、調整されているのがわかる。
<< ANOVA TABLE >>
== Adjusted by Greenhouse-Geisser's Epsilon ==
---------------------------------------------------------------
Source SS df MS F-ratio p-value
---------------------------------------------------------------
A 42.5633 1 42.5633 2.5404 0.1420 ns
s x A 167.5433 10 16.7543
---------------------------------------------------------------
B 205.1500 1.11 185.1848 113.6565 0.0000 ***
A x B 35.5000 1.11 32.0451 19.6676 0.0008 ***
s x A x B 18.0500 11.08 1.6293
---------------------------------------------------------------
Total 468.8067 47 9.9746
+p < .10, *p < .05, **p < .01, ***p < .001
Huynh-Feldt-Lecoutreの方法による結果はこちら。
<< ANOVA TABLE >>
== Adjusted by Huynh-Feldt-Lecoutre's Epsilon ==
---------------------------------------------------------------
Source SS df MS F-ratio p-value
---------------------------------------------------------------
A 42.5633 1 42.5633 2.5404 0.1420 ns
s x A 167.5433 10 16.7543
---------------------------------------------------------------
B 205.1500 1.15 179.0933 113.6565 0.0000 ***
A x B 35.5000 1.15 30.9910 19.6676 0.0007 ***
s x A x B 18.0500 11.45 1.5757
---------------------------------------------------------------
Total 468.8067 47 9.9746
+p < .10, *p < .05, **p < .01, ***p < .001
Greenhouse-Geisserの方法やHuynh-Feldt-Lecoutreの方法を適用するかどうかは、球面検定という等分散性の検定の結果を確認する。
p値は小さく、統計学的有意なので、調整を使った結果のほうがより適切である。
<< SPHERICITY INDICES >>
== Mendoza's Multisample Sphericity Test and Epsilons ==
-------------------------------------------------------------------------
Effect Lambda approx.Chi df p LB GG HF CM
-------------------------------------------------------------------------
B 0.0000 39.2530 11 0.0001 *** 0.3333 0.3693 0.3818 0.3405
-------------------------------------------------------------------------
LB = lower.bound, GG = Greenhouse-Geisser
HF = Huynh-Feldt-Lecoutre, CM = Chi-Muller
分割プロット分散分析をEZRで行う方法
EZRを使って、分割プロット分散分析を行ってみた。
データは上記のANOVA君に使ったデータだ。
CSVファイルに出力して使う。
write.csv(dat.AsB, "split-plot-design.csv", row.names=FALSE)
「ファイル」→「データのインポート」→「ファイルまたはクリップボード,URLからテキストデータを読み込む」で、CSVファイルをアクティブデータセットに読み込む。
「統計解析」→「連続変数の解析」→「対応のある2群以上の間の平均値の比較(反復[経時]測定分散分析)を選ぶ。
以下のように変数を選択する。
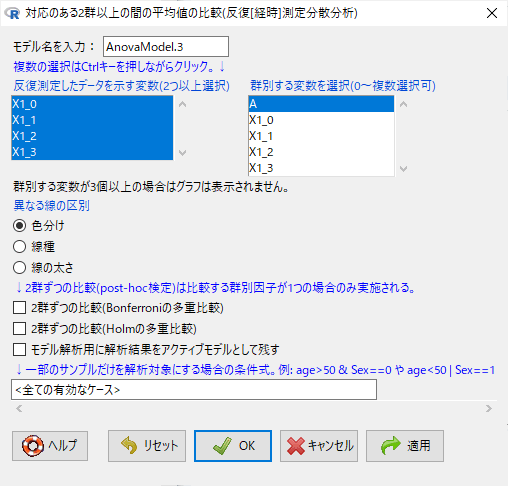
OKか適用をクリックすれば、グラフが表示され、計算結果が表示される。
グラフはこんな感じ。
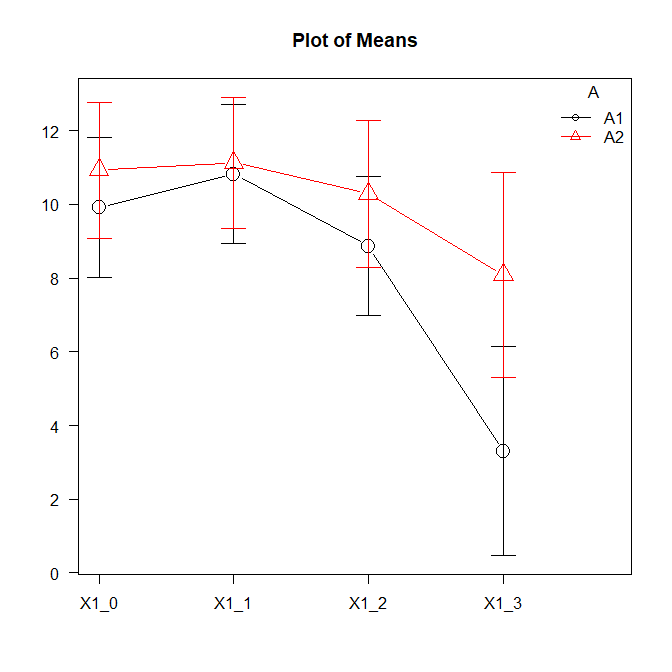
計算結果は以下の通り。
> summary(res, multivariate=FALSE)
Univariate Type III Repeated-Measures ANOVA Assuming Sphericity
Sum Sq num Df Error SS den Df F value Pr(>F)
(Intercept) 4033.3 1 167.54 10 240.7337 0.00000002526 ***
Factor1.A 42.6 1 167.54 10 2.5404 0.142
Time 205.1 3 18.05 30 113.6565 < 2.2e-16 ***
Factor1.A:Time 35.5 3 18.05 30 19.6676 0.00000030505 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Mauchly Tests for Sphericity
Test statistic p-value
Time 0.011005 0.00000026448
Factor1.A:Time 0.011005 0.00000026448
Greenhouse-Geisser and Huynh-Feldt Corrections
for Departure from Sphericity
GG eps Pr(>F[GG])
Time 0.36927 0.0000002613 ***
Factor1.A:Time 0.36927 0.0007995 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
HF eps Pr(>F[HF])
Time 0.3818309 0.0000001709083
Factor1.A:Time 0.3818309 0.0006813689172
Factor1.Aが上記のA(グループ)にあたり、Timeが上記のB(測定回)にあたる。
Mauchly(モークリー)の球面検定(Sphericity)が統計学的に有意で、等分散が仮定できないので、Greenhouse-Geisser(グリーンハウス-ガイサー)やHuynh-Feldt (Lecoutre)(フイン-フェルト-ルクトル)の修正後の結果を見るのがよい。
Greenhouse-Geisserはコンサバすぎるので、Huynh-Feldt-Lecoutreの方法がよい。
表示はHuynh-Feldtとしか書かれていないが、ANOVA君の結果と同じことから、Lecoutreが指摘した修正が適用されていると理解できる。
まとめ
分割プロット分散分析、群分けがある反復測定分散分析を R 、ANOVA 君及び EZR で行う方法を紹介した。
関連記事
Greenhouse-Geisserの方法及びHuynh-Feldtの方法

参考書籍
医学への統計学 8.5.3 経時的変動パターンの差に興味がある場合

新版はこちら。





コメント
コメント一覧 (3件)
[…] R で分割プロット分散分析を行う方法 分割プロット分散分析 split-plot design […]
[…] R で分割プロット分散分析を行う方法 分割プロット分散分析 split-plot design […]
[…] R で分割プロット分散分析を行う方法 分割プロット分散分析 split-plot design […]