平均値の差のメタ解析のやり方を解説。
メタ解析のやり方解説のためのサンプルデータ
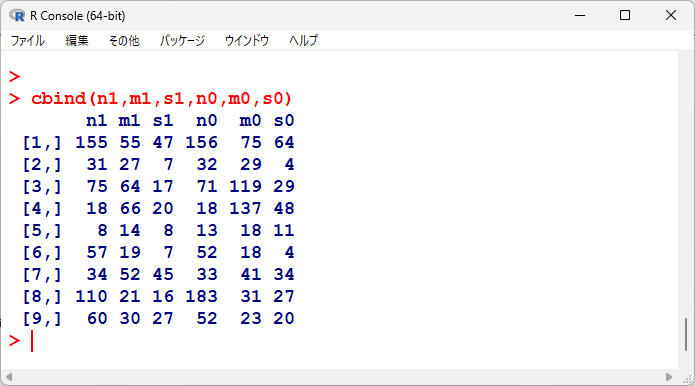
メタ解析のやり方を解説するためのデータは以下の通り。
mが平均、sが標準偏差、nがサンプルサイズ。
n1 <- c(155,31,75,18,8,57,34,110,60)
m1 <- c(55.0,27.0,64.0,66.0,14.0,19.0,52.0,21.0,30.0)
s1 <- c(47.0,7.0,17.0,20.0,8.0,7.0,45.0,16.0,27.0)
n0 <- c(156,32,71,18,13,52,33,183,52)
m0 <- c(75.0,29.0,119.0,137.0,18.0,18.0,41.0,31.0,23.0)
s0 <- c(64.0,4.0,29.0,48.0,11.0,4.0,34.0,27.0,20.0)
並べてみると以下の通り。
それぞれの試験の平均値の差と95%信頼区間
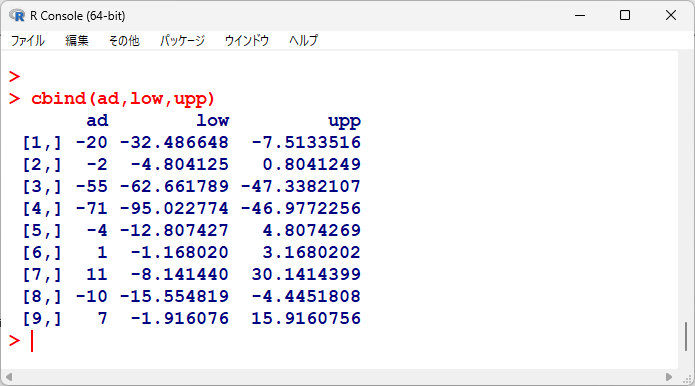
各試験の平均値の差を計算し、95%信頼区間を計算する。
# ---- difference in means -----
ad <- m1-m0
s <- ((n1-1)*s1^2+(n0-1)*s0^2)/(n1-1+n0-1)
se <- sqrt((1/n1+1/n0)*s)
tn <- n1+n0
low <- ad-1.96*se
upp <- ad+1.96*se
平均値の差と95%信頼区間の一覧を出力してみると、以下のようになる。
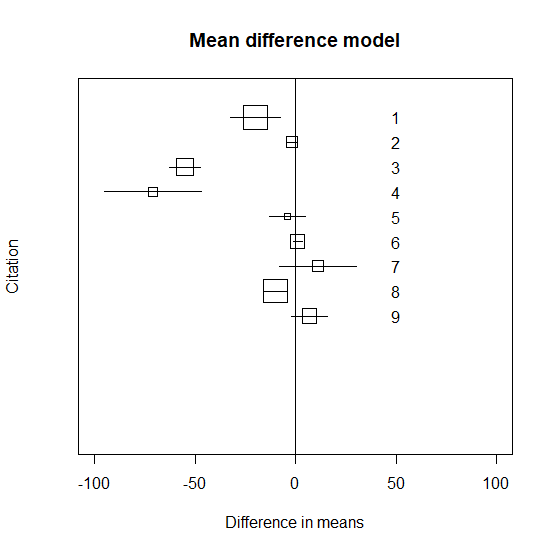
それぞれの試験の平均値の差と95%信頼区間のグラフ化
各試験の平均値の差と95%信頼区間をグラフに書いてみる。
# ----- individual graph -----
k <- length(n1)
id <- k:1
plot(ad, id, ylim=c(-4,10), pch=" ",
xlim=c(-100,100), yaxt="n",
ylab="Citation", xlab="Difference in means")
title(main=" Mean difference model ")
symbols(ad, id, squares=sqrt(tn), add=T, inches=0.25)
abline(v=0)
for (i in 1:k){
j <- k-i+1
x <- c(low[i], upp[i])
y <- c(j, j)
lines(x, y, type="l")
text(50, i, j)
}
メタ解析1 固定効果の計算
固定効果とは、研究の結果が似ていると思えるときに使える方法。
メタ解析には、まず固定効果で統合する。
# ----- fixed effects -----
w <- 1/se/se
sw <- sum(w)
adm <- sum(ad*w)/sw
adl <- adm-1.96*sqrt(1/sw)
adu <- adm+1.96*sqrt(1/sw)
q1 <- sum(w*(ad-adm)^2)
df1 <- k-1
pval1 <- 1-pchisq(q1, df1)
q2 <- adm^2*sw
df2 <- 1
pval2 <- 1-pchisq(q2, df2)
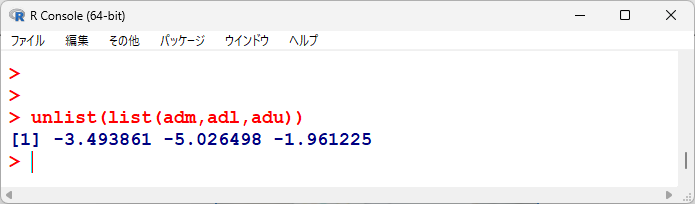
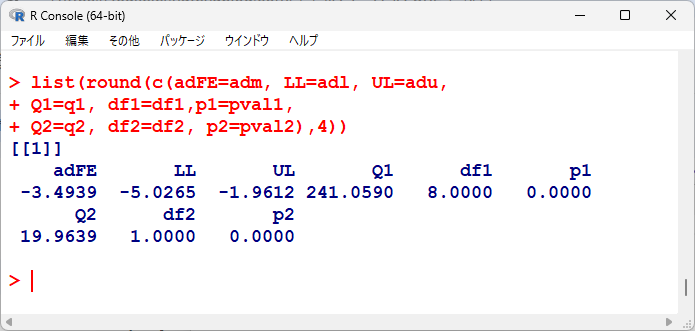
統合平均値差、95%信頼区間下限、上限を並べてみると以下の通り。
均質性の検定、有意性の検定の各p値を並べてみると以下の通り。
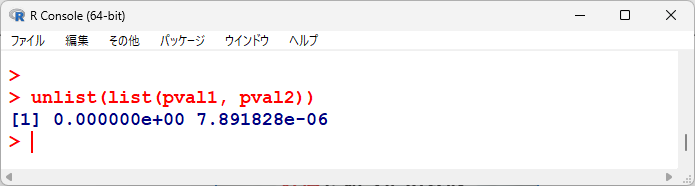
統計学的有意であるが、均質性の検定に関しても有意、つまり異質性ありなので、変量効果で統合するほうがよさそうである。
メタ解析2 変量効果の計算(DerSimonian-Lairdの方法)
変量効果とは、研究同士に無視できない違い(異質性)があるときに使う方法。
# ----- random effects -----
tau2 <- (q1-(k-1))/(sw-sum(w*w)/sw)
tau2 <- max(0, tau2)
wx <- 1/(tau2+se*se)
swx <- sum(wx)
admx <- sum(ad*wx)/swx
adxl <- admx-1.96*sqrt(1/swx)
adxu <- admx+1.96*sqrt(1/swx)
qx2 <- admx^2*swx
pvalx2 <- 1-pchisq(qx2, df2)
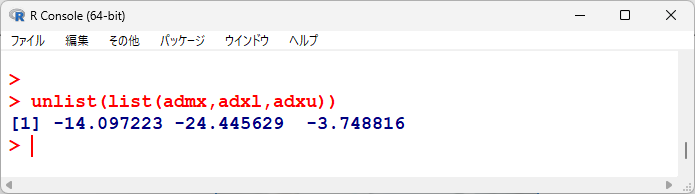
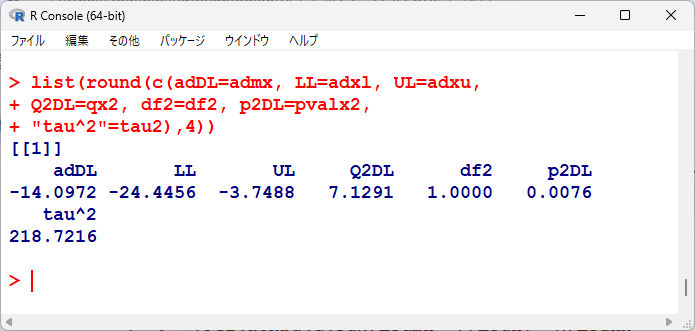
変量効果による統合点推定値、95%信頼区間を表示すると以下のとおりである。
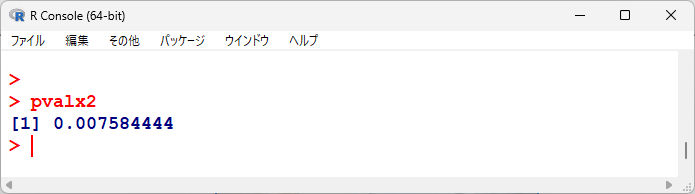
有意性の検定結果は以下のとおりである。
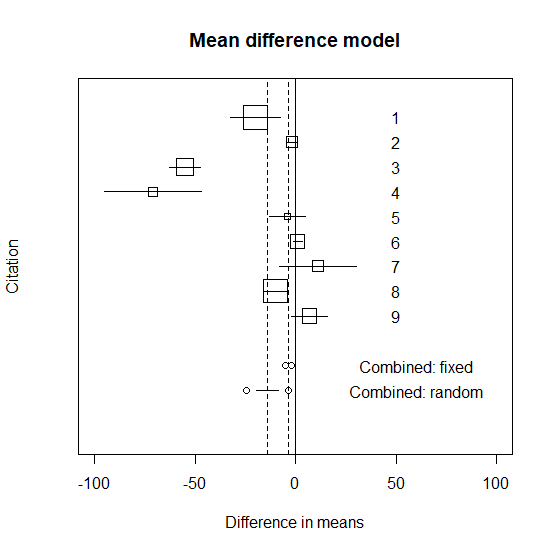
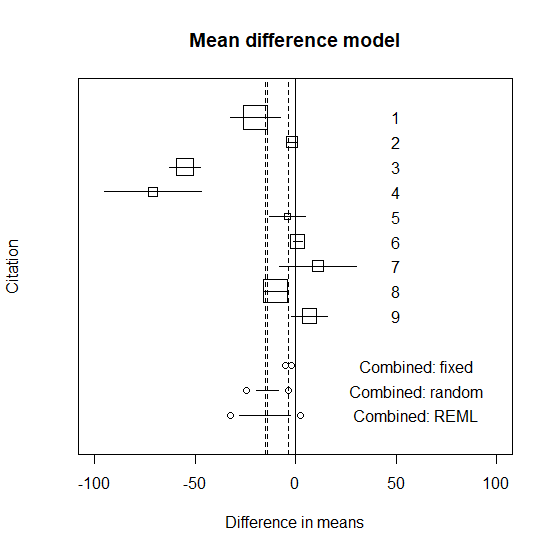
メタ解析のグラフに統合平均値と95%信頼区間を書き入れる
先ほどのグラフに統合平均値と95%信頼区間を書き入れる。
# ----- graph -----
x <- c(adl, adu)
y <- c(-1, -1)
lines(x, y, type="b")
x <- c(adxl, adxu)
y <- c(-2, -2)
lines(x, y, type="b")
abline(v=c(adm, admx), lty=2)
text(60, -1, "Combined: fixed")
text(60, -2, "Combined: random")
メタ解析の結果を出力する
ここまで、途中途中で結果を出力してきたが、以下にまとめて出力する。
adFE, LL, ULは、固定効果の統合平均値と95%信頼区間、Q1, df1, p1は均質性の検定(検定統計量、自由度、P値)、Q2, df2, p2は有意性の検定(検定統計量、自由度、P値)である。
adDL, LL, ULは変量効果の統合平均値と95%信頼区間、Q2DL, df2, p2DL は変量効果の有意性の検定、tau2は $ \tau^2 $ の推定値である。
メタ解析をより柔軟な変量モデル―制限付き最尤推定量(REML)で行う
変量モデルのDerSimonian-Lairdの方法は簡便な方法だが、より柔軟で理論的に自然な方法が制限付き最尤推定量(REML, restricted maximum likelihood estimator)だ。
現代はPCを使って反復計算が簡単にできるようになったので、REMLの適用が望ましい。
Newton-Raphson法を用いて繰り返し収束計算で $ \tau^2 $ の推定値を求めている。
# ----- REML method -----
intau <- tau2
tau <- intau
#
nrep <- 10
newt <- 1:nrep
for (i in 1:nrep){
wb <- 1/(tau+se*se)
admb <- sum(ad*wb)/sum(wb)
qf <- k/(k-1)*(ad-admb)^2-se*se
dkx <- (-1*sum(ad*wb*wb)+admb*sum(wb*wb))/sum(wb)
qf2 <- -2*k/(k-1)*(ad-admb)*dkx
h <- sum(wb*wb*(qf-tau))
dh <- sum(-2*wb*wb*wb*(qf-tau)+wb*wb*(qf2-1))
newt[i] <- tau - h/dh
rel <- abs((newt[i]-tau)/tau)
tau <- newt[i]
}
# ----- 統合値の計算 有意性の検定 -----
wg <- 1/(tau+se*se)
swg <- sum(wg)
adRM <- sum(ad*wg)/swg
adRMl <- adRM-1.96*sqrt(1/swg)
adRMu <- adRM+1.96*sqrt(1/swg)
qx2RM <- adRM^2*swg
pvalx2RM <- 1-pchisq(qx2RM, df2)
tau2 <- tau
# ----- 統合値をグラフに書き入れる -----
x <- c(adRMl, adRMu)
y <- c(-3, -3)
lines(x, y, type="b")
abline(v=adRM, lty=2)
text(60, -3, "Combined: REML")
REMLの結果をグラフに足すと以下のようになる。
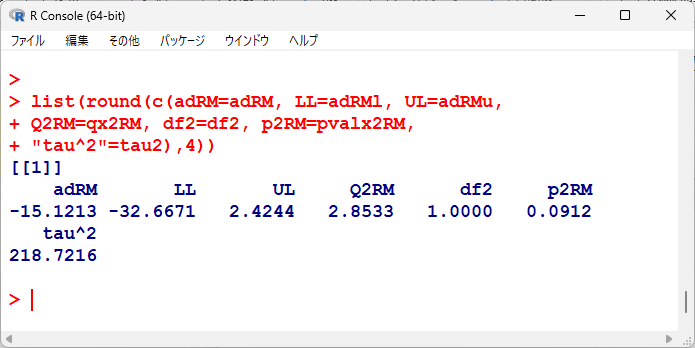
計算結果は、以下の通り。
DerSimonian-Lairdの方法よりもさらに95%信頼区間が広がっていて、統計学的有意でなくなっている。
メタ解析を metafor パッケージを使って行う方法
Rのmetafor パッケージを使うと簡単にメタ解析ができる。
インストールしたあと、library() で呼び出しておく。
install.packages("metafor") #1回のみ
library(metafor) #使うときに
計算は以下のように行う。
ポイントは、escalc()関数とrma.uni()関数である。
# データ再掲
n1 <- c(155,31,75,18,8,57,34,110,60)
m1 <- c(55.0,27.0,64.0,66.0,14.0,19.0,52.0,21.0,30.0)
s1 <- c(47.0,7.0,17.0,20.0,8.0,7.0,45.0,16.0,27.0)
n0 <- c(156,32,71,18,13,52,33,183,52)
m0 <- c(75.0,29.0,119.0,137.0,18.0,18.0,41.0,31.0,23.0)
s0 <- c(64.0,4.0,29.0,48.0,11.0,4.0,34.0,27.0,20.0)
# データフレームに変換
dat <- data.frame(m1, s1, n1, m0, s0, n0)
# 推定値と分散の計算
dat.escalc <- escalc(measure="MD", vtype="HO",
m1i=m1, sd1i=s1, n1i=n1,
m2i=m0, sd2i=s0, n2i=n0,
data=dat)
# 統合値の計算
res.reml <- rma.uni(yi, vi, method="REML",
data=dat.escalc)
summary(res.reml)
escalc()関数で、measure=”MD”としているところが平均値の差 (mean difference) の統合に向けた計算を意味している。
vtype=”HO” は、分散均一性 homoscedasticity を想定するための指定だ。
デフォルトでは、分散均一性を想定しない方法 vtype=”LS” を用いている。
通常では、指定せず、デフォルトのままでよい。
今回の記事では、標準誤差を計算する方法が分散均一性を仮定しているので、それに合わせるために指定した。
各試験における2群の標準偏差を平均値の差の標準偏差及び標準誤差に変換する際に、分散均一性を想定した計算に仕方をしているところに由来する。
統合結果は以下の通りだ。
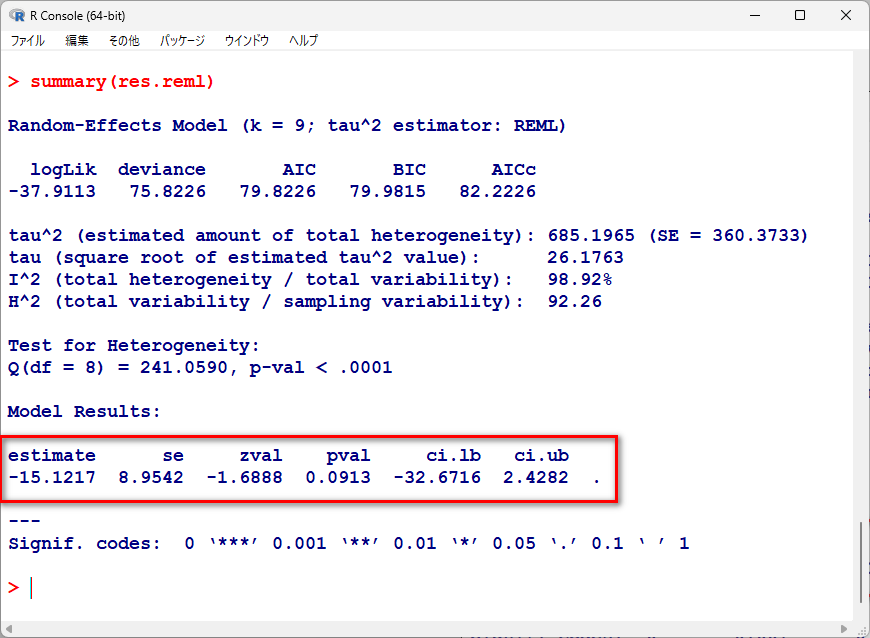
上記のREML法での結果と若干異なるものの、ほぼ同様の点推定値、95%信頼区間、p値であると言える。
まとめ
試験のエンドポイントが平均値の差の検定である試験を統合する方法を下記引用書籍に沿って実施してみた。
図を見ると明らかに試験同士が大きく異なっていて、無視できない差があると理解できる。
試験同士に無視できない差がある場合は、変量効果モデル(REML)を使うことが望ましい。
引用書籍
丹後敏郎著 メタ・アナリシス入門
3.2 平均値と標準偏差

新版もある。






コメント
コメント一覧 (2件)
[…] R で平均値の差のメタ解析を行う方法 平均値の差のメタ解析のやり方を解説。 […]
[…] R で平均値の差のメタ解析を行う方法 平均値の差のメタ解析のやり方を解説。 […]