この記事では「ノモグラムとは?医療分野でも使われる予測モデルを可視化するツール」としてお伝えします。
- ノモグラムとは?医療で使われる診断ツール
- ノモグラムの具体的な使い方
- ノモグラムの作り方!EZRでは作れないけどRのrmsパッケージで作成可能
をお伝えしますので、今日からでもRでノモグラムを作成することができるようになると思います!
ノモグラムとは?医療で使われる診断ツール
ノモグラムは、ある関数(予測モデルや診断モデルなど)によって計算された予測確率のアウトプットを可視化するためのツールです。
実際のモノを見た方がイメージが湧きやすいと思いますので、結論から言えば以下のようなモノです。
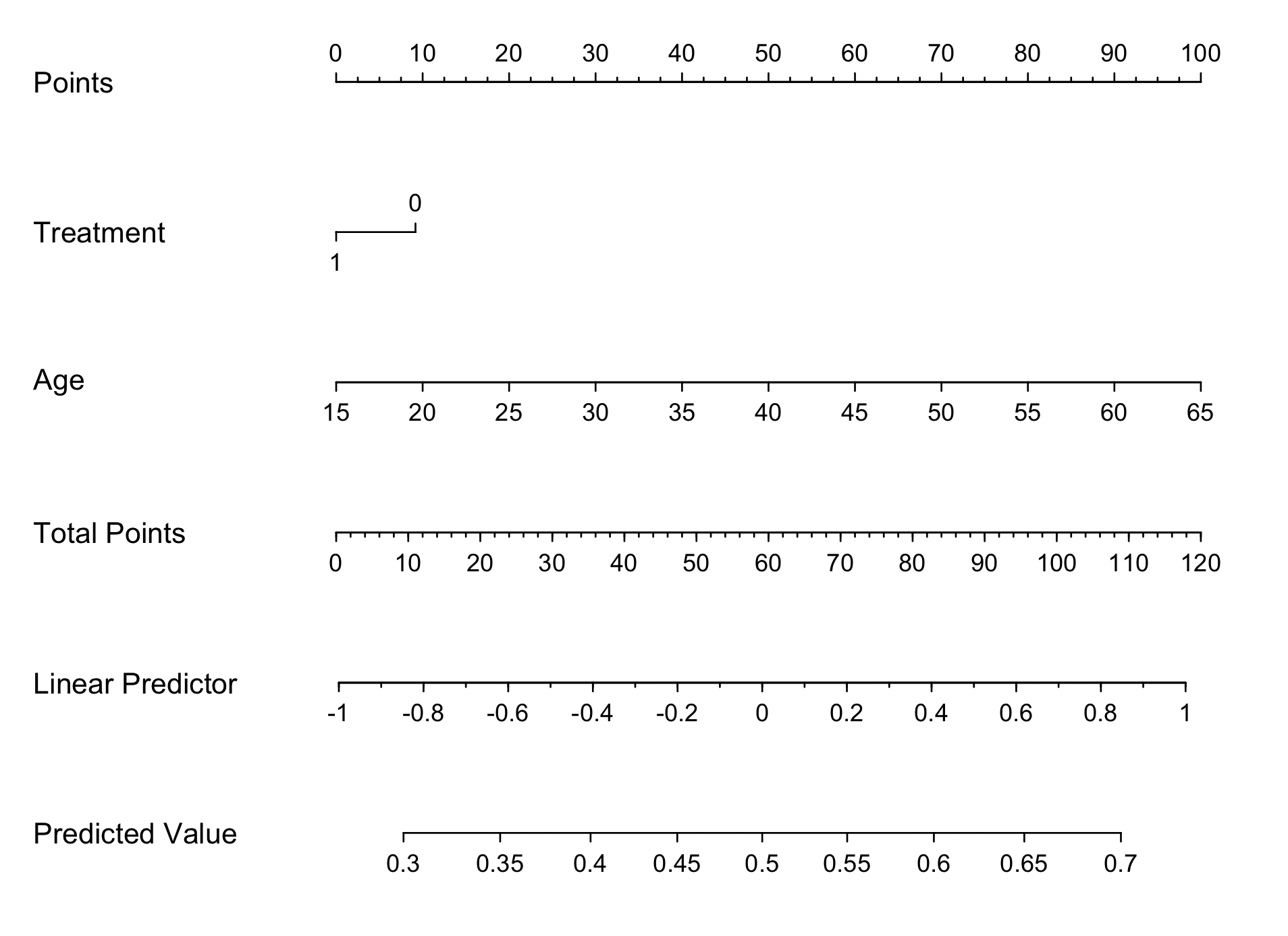
ノモグラムの目的は「予測確率を計算する」ですが、その目的であれば、予測モデルの数式から計算した方が正確です。
確かに、予測モデルの数式から予測確率を計算することも可能なのですが、ノモグラムを使うと大まかな予測確率を可視化しながら計算できるというメリットがあります。
つまり、Cox回帰やロジスティック回帰を知らなくても直感的に計算が可能です。
「Cox回帰やロジスティック回帰を知らない」ということ自体は、あまり褒められたものではありませんが。。
そして、ノモグラムであれば計算過程も可視化できますし、各説明変数の重みも視覚的に分かりやすいです。
上記で示したノモグラムをみると、「Treatment」という因子よりも「Age」という因子の方が予測確率に寄与していそうなことがわかります。
ノモグラムは医療現場でもがんの分野でよく使われている印象
ノモグラムは、医療現場ではがんの分野でよく使われている印象があります。
例えば、大腸がん研究会の「結腸癌術後予後予測ノモグラム」というものがあります。
>>大腸がん研究会の「結腸癌術後予後予測ノモグラム」はこちらから
入力画面で、現在の年齢や各種臨床検査の値を入力していきます。
すると、3年後の予測生存割合、5年後の予測生存割合が出力されます。
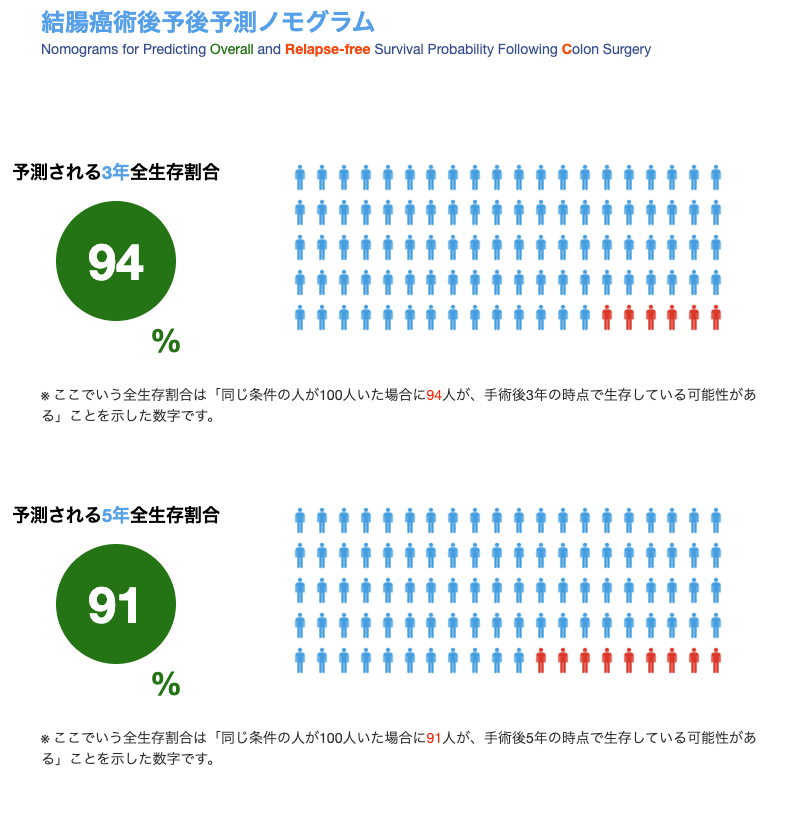
このような感じで、予測モデルを「可視化」できることがノモグラムの何よりも強みになっています。
ノモグラムの具体的な使い方
ノモグラムが何なのかが把握できたところで、具体的に使い方を見ていきましょう。
この記事では、こちらの論文にあったノモグラムを使ってみます。

結論から言えば、この論文で作成されたノモグラムはこちらです。
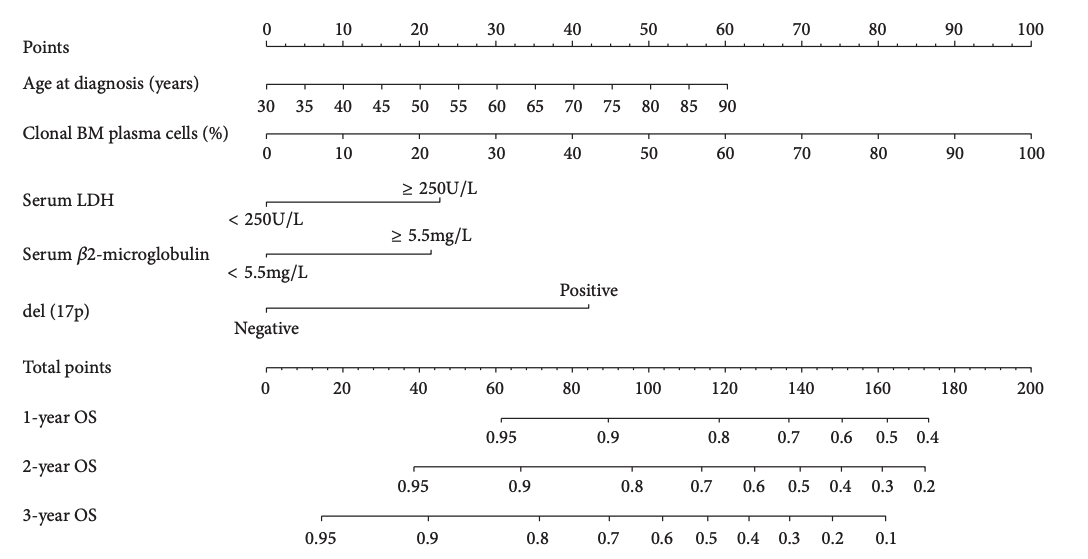
このノモグラムからわかることは、「年齢」「Clonal BM plasma cell」「LDH」「β2ーmicroglobulin」「del(17p)」の5つの値があれば、1年後・2年後・3年後の生存割合を知ることができるということ。
仮に、下記のような方がいたとして、この症例の生存率を予測してみます。
- 年齢:40歳
- Clonal BM plasma cell:20%
- LDH:<250U/L
- β2-microglobulin:>=5.5mg/L
- del(17p):Positive
まずは、この症例の総合得点を計算します。(図の赤字部分)
この症例は、0+10+20+22+42=94点を持つことがわかります。
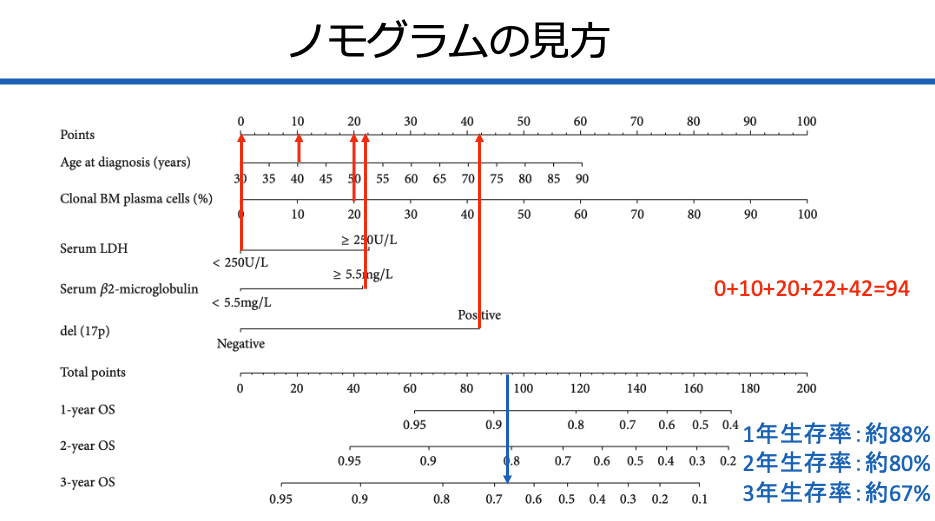
次に、総合得点と対比する生存率を確認します(図の青字部分)。
すると、1年生存率:約88%、2年生存率:約80%、3年生存率:約67%、という予測になっていることがわかります。
以上が、ノモグラムの具体的な使い方です。
ノモグラムの作り方!EZRでは作れないけどRのrmsパッケージで作成可能
このノモグラムは、結構簡単に作ることができます。
残念ながらEZRのクリック操作だけでは作ることができないのですが、EZRの裏で動いているRのrmsパッケージというものがあれば作成可能。
そのためここからは、Rのrmsパッケージを使って
- Cox比例ハザードモデルでのノモグラム作成方法
- ロジスティック回帰でのノモグラム作成方法
をお伝えします。
データは自治医科大学さんから提供されている「Survival.csv」を使っていきます。
前提として、rmsパッケージをインストールしていることを想定しています。
パッケージのインストール方法は「r パッケージインストール」とかで検索してみてください。
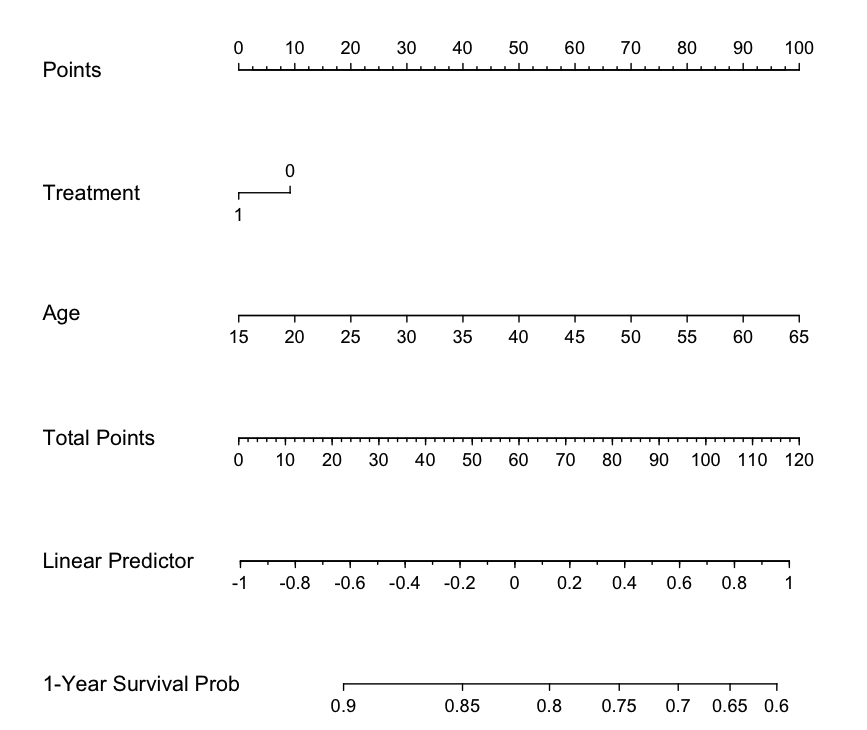
Cox比例ハザードモデルでのノモグラム
状況としては、「前治療の有無(Treatment)」「年齢(Age)」の2つで生存率を予測するノモグラムを作りたいとします。
そして結論から言えば、下記のようなRのプログラムを実行することで作成可能です。
(例えば、1年時点の生存率を予測する場合)
# ノモグラムの作成
library(rms)
dat <- read.csv("Survival.csv")
cox.mod<- cph(Surv(DaysToOS, OS > 0) ~ Treatment + Age, data = dat, x=TRUE,y=TRUE,surv=TRUE)
cox.mod
dd <- datadist(dat)
options(datadist="dd")
cox_nomogram <- nomogram(cox.mod, lp=TRUE, fun= list(function(x) surv(365,x)), funlabel="1-Year Survival Prob")
plot(cox_nomogram)
すると、下記のようなノモグラムが作成されます。
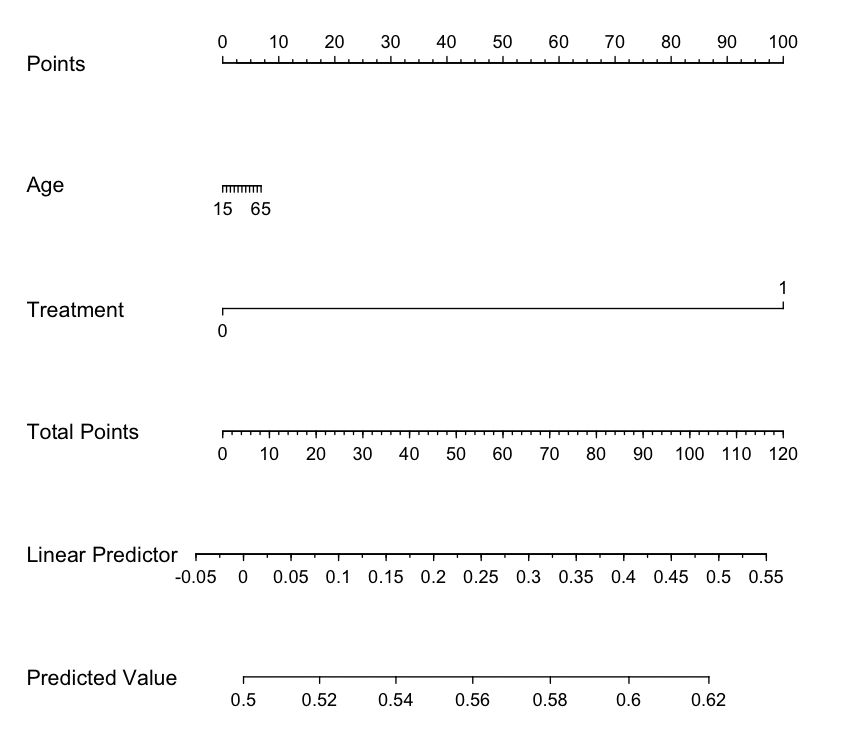
ロジスティック回帰でのノモグラム
「前治療の有無(Treatment)」「年齢(Age)」の2つで性別を予測するノモグラムを作りたいとします。
(性別を予測する、という状況は現実的にはあり得ないですが。。)
# ノモグラムの作成
library(rms)
dat <- read.csv("Survival.csv")
log.result <- lrm(Sex ~ Age + Treatment, data=dat)
log.result
dd <- datadist(dat)
options(datadist="dd")
log_nomogram <- nomogram(log.result, fun = plogis)
plot(log_nomogram)
すると、下記のようなノモグラムが作成されます。
Rでノモグラム作成の際の修正Tips
Rでノモグラム作成することができました。
ただ、デフォルトで出力されるノモグラムの表現を修正したい、という場合もあると思います。
そのため、修正のTipsをお伝えします。
具体的には下記2点です。
- linear predictorを消す
- Predicted Valueの名称を変更
ノモグラムのlinear predictorを消す
ノモグラムのlinear predictorを消すためには、nomogram関数の中で「lp=F」を追記します。
具体的には、以下のようなプログラムに。
# ノモグラムの作成
library(rms)
dat <- read.csv("Survival.csv")
log.result <- lrm(Sex ~ Age + Treatment, data=dat)
log.result
dd <- datadist(dat)
options(datadist="dd")
log_nomogram <- nomogram(log.result, fun = plogis, lp=F)
plot(log_nomogram)
すると、linear predictorが消えることがわかります。
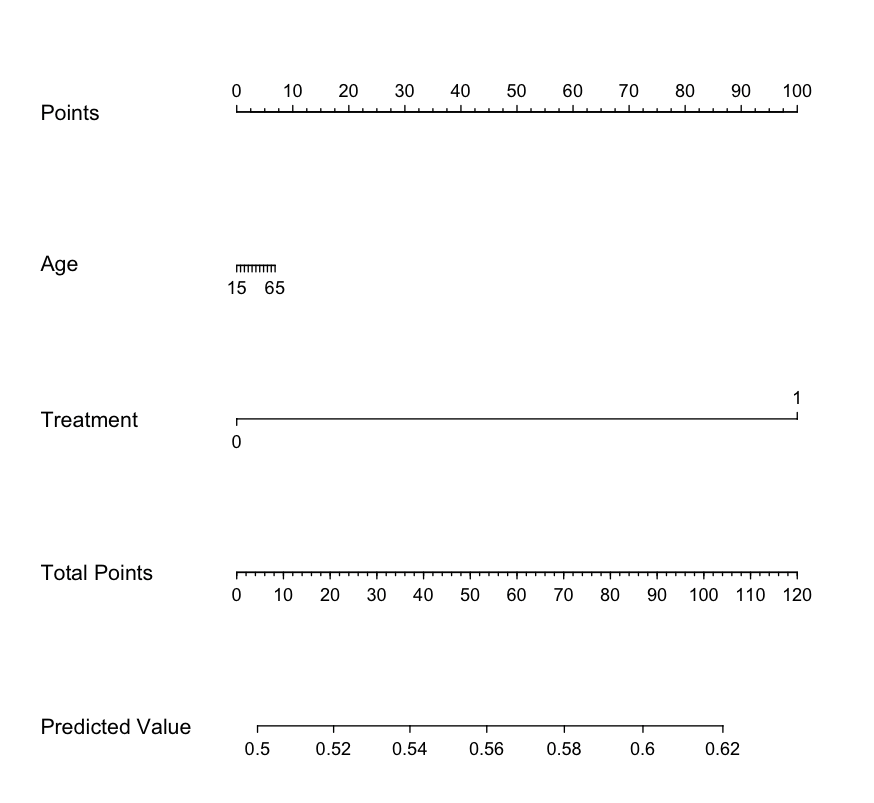
ノモグラムのPredicted valueの名称を変更
ノモグラムのPredicted valueの名称を変更するためには、nomogram関数の中で「funlabel=”修正したい名前”」を追記します。
具体的には、下記のようなプログラムです。(Yosokuという名前に修正する例です。)
# ノモグラムの作成
library(rms)
dat <- read.csv("Survival.csv")
log.result <- lrm(Sex ~ Age + Treatment, data=dat)
log.result
dd <- datadist(dat)
options(datadist="dd")
log_nomogram <- nomogram(log.result, fun = plogis, lp=F, funlabel="Yosoku")
plot(log_nomogram)
すると、Yosokuという名前になることがわかります。
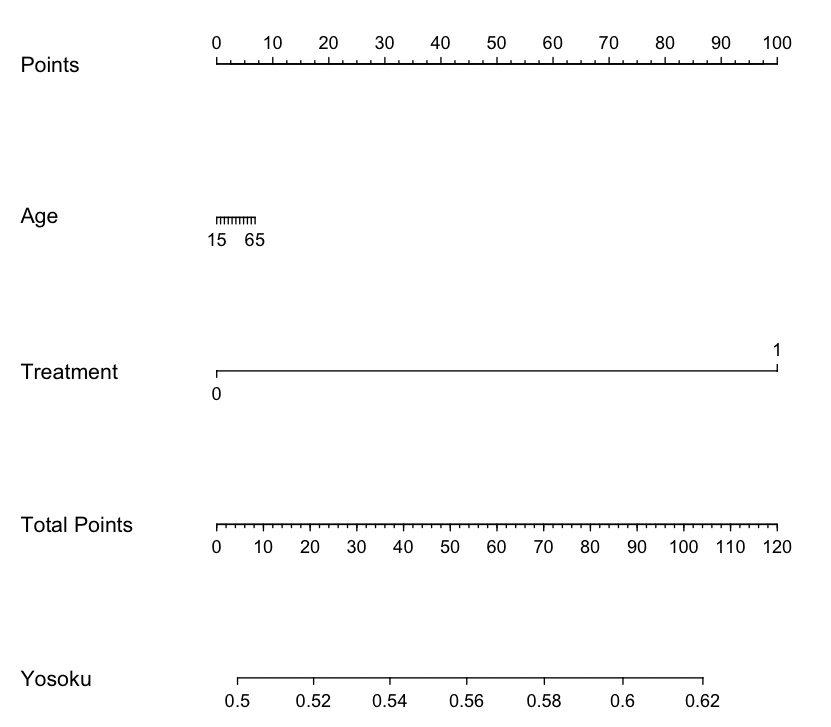
まとめ
いかがでしたか?
この記事では「ノモグラムとは?医療分野でも使われる予測モデルを可視化するツール」としてお伝えしました。
- ノモグラムとは?医療で使われる診断ツール
- ノモグラムの具体的な使い方
- ノモグラムの作り方!EZRでは作れないけどRのrmsパッケージで作成可能
をご理解いただけれたのなら幸いです!
こちらの内容は動画でも解説しておりますので、併せてご確認くださいませ。













コメント